SeekSoul Tools v1.2.2
SeekSoulTools 是寻因生物开发的一套处理单细胞转录组数据的软件,目前该软件包含四个模块:
1.rna模块;用于识别细胞标签barcode,比对定量,得到可用于下游分析的细胞表达矩阵,之后进行细胞聚类和差异分析,该模块不仅支持SeekOne®系列试剂盒产出数据,还 可通过对barcode的描述,支持多种自定义设计结构;
2.fast模块:该模块专门针对SeekOne® DD单细胞全序列转录组试剂盒产出的数据,用于对数据进行barcode提取,双端reads比对,定量,以及全序列数据特有的指标统计。
3.vdj模块:该模块用于专门针对SeekOne® DD单细胞免疫分析试剂盒产出的数据,用于组装、过滤和注释免疫受体;
4.utils模块:该模块包含其他小工具。
软件下载
SeekSoul Tools v1.2.2
Download-SeekSoulTools - md5: af52b5d936b60c740b239301f2026aba
wget下载方式
mkdir seeksoultools.1.2.2
cd seeksoultools.1.2.2
wget -c -O seeksoultools.1.2.2.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/seeksoultools/seeksoultools.1.2.2.tar.gz"curl下载方式
mkdir seeksoultools.1.2.2
cd seeksoultools.1.2.2
curl -C - -o seeksoultools.1.2.2.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/seeksoultools/seeksoultools.1.2.2.tar.gz"软件安装
IMPORTANT
安装前请确保已正确配置conda环境,避免与其他生信软件环境冲突。
安装:
# 解压
tar zxf seeksoultools.1.2.2.tar.gz
# 设置环境变量
export PATH=`pwd`:$PATH
echo "export PATH=$(pwd):\$PATH" >> ~/.bashrc
# 初始化和安装验证,初次执行所需时间稍长
./seeksoultools --versionTIP
建议将PATH添加到~/.bashrc,便于后续命令行直接调用。
NOTE
若出现"command not found"或依赖报错,请检查conda环境激活及PATH设置。
使用说明
rna模块
数据准备
测试数据下载
测试数据 - md5: 3d15fcfdefc0722735d726f40ec4e324(物种:人)
wget下载方式
wget -c -O demo_dd.tar "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/demodata/demo_dd.tar"
# decompress
tar xf demo_dd.tarcurl下载方式
curl -C - -o demo_dd.tar "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/demodata/demo_dd.tar"
# decompress
tar xf demo_dd.tar参考基因组下载和构建
Download-human-reference-GRCh38 - md5: 5473213ae62ebf35326a85c8fba6cc42
Download-mouse-reference-mm10 - md5: 5c7c63701ffd7bb5e6b2b9c2b650e3c2
wget下载方式
wget -c -O GRCh38.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/reference/GRCh38.tar.gz"
# decompress
tar -zxvf GRCh38.tar.gz
wget -c -O mm10.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/reference/mm10_ensemble_102.tar.gz"
tar -zxvf mm10.tar.gzcurl下载方式
curl -C - -o GRCh38.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/reference/GRCh38.tar.gz"
# decompress
tar -zxvf GRCh38.tar.gz
curl -C - -o mm10.tar.gz "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/reference/mm10_ensemble_102.tar.gz"
tar -zxvf mm10.tar.gz参考基因组的构建可以参考如何构建参考基因组?
CAUTION
下载和解压参考基因组时,请确保磁盘空间充足,避免文件损坏导致后续分析失败。
运行
运行示例
示例1:基本用法
为分析设置必要的配置文件,包括样本数据的路径、试剂类型、基因组索引、GTF等。使用以下命令运行SeekSoulTools:
seeksoultools rna run \
--fq1 /path/to/demo_dd/demo_dd_S39_L001_R1_001.fastq.gz \
--fq2 /path/to/demo_dd/demo_dd_S39_L001_R2_001.fastq.gz \
--samplename demo_dd \
--genomeDir /path/to/GRCh38/star \
--gtf /path/to/GRCh38/genes/genes.gtf \
--chemistry DDV2 \
--core 4 \
--include-introns示例2:指定其他版本STAR进行分析
为了在分析中使用特定版本的STAR,并确保与该版本生成的–genomeDir兼容,你可以使用以下命令运行SeekSoulTools,并指定所需版本的STAR的路径:
seeksoultools rna run \
--fq1 /path/to/demo_dd/demo_dd_S39_L001_R1_001.fastq.gz \
--fq2 /path/to/demo_dd/demo_dd_S39_L001_R2_001.fastq.gz \
--samplename demo_dd \
--genomeDir /path/to/GRCh38/star \
--gtf /path/to/GRCh38/genes/genes.gtf \
--chemistry DDV2 \
--core 4 \
--include-introns \
--star_path /path/to/cellranger-5.0.0/lib/bin/STARNOTE
--star_path参数需与参考基因组索引版本严格对应,否则会导致比对失败。
示例3:一个样本有多组fastq数据
如果一个样本有多个FASTQ数据集,你可以在运行SeekSoulTools时提供与该样本相关的所有FASTQ文件的路径。以下是一个示例命令:
seeksoultools rna run \
--fq1 /path/to/demo_dd_S39_L001_R1_001.fastq.gz \
--fq1 /path/to/demo_dd_S39_L002_R1_001.fastq.gz \
--fq2 /path/to/demo_dd_S39_L001_R2_001.fastq.gz \
--fq2 /path/to/demo_dd_S39_L002_R2_001.fastq.gz \
--samplename demo \
--genomeDir /path/to/GRCh38/star \
--gtf /path/to/GRCh38/genes/genes.gtf \
--chemistry DDV2 \
--core 4 \
--include-introns示例4:自定义R1结构
要自定义 Read 1 (R1) FASTQ文件的结构,可以使用以下命令:
seeksoultools rna run \
--fq1 /path/to/demo_dd_S39_L001_R1_001.fastq.gz \
--fq2 /path/to/demo_dd_S39_L001_R2_001.fastq.gz \
--samplename demo \
--genomeDir /path/to/GRCh38/star \
--gtf /path/to/GRCh38/genes/genes.gtf \
--barcode /path/to/utils/CLS1.txt \
--barcode /path/to/utils/CLS2.txt \
--barcode /path/to/utils/CLS3.txt \
--linker /path/to/utils/Linker1.txt \
--linker /path/to/utils/Linker2.txt \
--structure B9L12B9L13B9U8 \
--core 4 \
--include-intronsB9L12B9L13B9U8表示read1的结构为:9个碱基barcode+12个碱基linker+9个碱基barcode+13个碱基linker+9个碱基barcode+9碱基UMI,整体cellbarcode有3段,共27个碱基(9*3),UMI为8个碱基- 使用
--barcode依次指定3段barcode,--linker依次指定2段linker。
TIP
自定义结构时,建议先用小样本测试结构参数,确保barcode/linker/UMI提取无误。
软件参数说明
| 参数 | 参数说明 |
|---|---|
| --fq1 | R1 fastq数据路径。 |
| --fq2 | R2 fastq数据路径。 |
| --samplename | 样本名称,会在outdir目录下创建以样本名称命名的目录。仅支持数字,字母和下划线。 |
| --outdir | 结果输出目录。默认值:./。 |
| --genomeDir | STAR构建的参考基因组路径, 版本需要与SeekSoulTools使用的STAR一致。 |
| --gtf | 相应物种的gtf路径。 |
| --core | 分析使用的线程数。 |
| --chemistry | 试剂类型,每种对应一组--shift、--pattern、 --structure、--barcode和--sc5p的组合,可选值:DDV2,DD5V1,MM,MM-D;DDV2 对应SeekOne(R) DD单细胞3'转录组试剂盒; DD5V1 对应SeekOne(R) DD单细胞5'转录组试剂盒; MM 对应SeekOne(R) MM单细胞转录组试剂盒; MM-D 对应SeekOne(R) MM大孔径高通量转录组试剂盒 |
| --skip_misB | barcode不允许碱基错配,默认允许一个碱基错配。 |
| --skip_misL | linker不允许碱基错配,默认允许一个碱基错配。 |
| --skip_multi | 舍弃能纠错为多个白名单barocde的reads,默认纠错为比例最高的barcode。 |
| --expectNum | 预估的捕获细胞数目。 |
| --forceCell | 当正常分析得到的细胞数⽬不理想时,选⽤此参数,后⾯加⼀个预期的数值N,SeekSoulTools软件会按照UMI从⾼到低取前N个细胞。 |
| --include-introns | 不启用时,只会选择exon reads⽤于定量;启用时,intron reads也会⽤于定量。 |
| --star_path | 指定其他版本的STAR路径进行比对,版本需要与--genomeDir版本兼容,默认的--star_path为环境下的STAR。 |
WARNING
参数设置不当(如chemistry、genomeDir、gtf等)会直接导致分析失败或结果异常,请务必核对参数含义和输入路径。
结果文件说明
以下是输出目录结构:每一行代表一个文件或文件夹,用 "├──" 表示,数字表示三个重要的输出文件。
./
├── demo_report.html 1
├── demo_summary.csv 2
├── demo_summary.json
├── step1
│ └──demo_2.fq.gz
├── step2
│ ├── featureCounts
│ │ └── demo_SortedByName.bam
│ └── STAR
│ ├── demo_Log.final.out
│ └── demo_SortedByCoordinate.bam
├── step3
│ ├── filtered_feature_bc_matrix 3
│ └── raw_feature_bc_matrix
└──step4
├── FeatureScatter.png
├── FindAllMarkers.xls
├── mito_quantile.xls
├── nCount_quantile.xls
├── nFeature_quantile.xls
├── resolution.xls
├── top10_heatmap.png
├── tsne.png
├── tsne_umi.png
├── tsne_umi.xls
├── umap.png
└── VlnPlot.png- 样本的html报告
- 样本的csv格式质控信息
- 算法判定是细胞的稀疏矩阵
NOTE
若输出目录缺失关键文件,请优先检查上游步骤是否报错,或参数设置是否正确。
处理过程
step1: barcode/UMI提取
SeekSoulTools根据不同的Read1的结构设计和参数对barcode/UMI进行提取和处理,对Read1和Read2进行过滤,输出新的fastq文件。
结构设计和描述
barcode和UMI描述 以字母和数字描述Read1的基本结构,字母描述碱基含义,数字描述碱基长度。
B: barcode部分碱基
L: linker部分碱基
U: UMI部分碱基
X: 其他任意碱基,用于占位以下面两种Read1结构为例:
B8L8B8L10B8U8:
B17U12:

错位设计的Anchor定位 MM设计下,为了增加Linker部分在测序时碱基均衡性,加入了1-4 bp的移码碱基,即anchor,anchor决定了barcode的起始位置。

数据流程
确定anchor:
对于Read1有错位设计的数据(MM试剂产出的数据),SeekSoulTools尝试在Read1序列前7个碱基中寻找anchor序列,以确定后续barcode等的起始。若没找到anchor序列,此条read以及对应的R2被认为是无效read。
Barcode和Linker矫正:
在确定barcode的起始后,根据结构设计,取出相应序列。当取出的barcode序列在白名单中时,我们认为它是有效barcode,计入有效barcode的reads数量;当barcode不在白名单中时,我们认为它是无效barcode。
测序过程中,有一定几率发生测序错误。在提供有白名单的情况下,SeekSoulTools可以尝试barcode矫正。在启用矫正时,当无效barcode一个碱基错配(一个hamming distance)的序列存在于白名单中:
- 只有唯一一个序列存在于白名单中:我们将这个无效barcode矫正为白名单中barcode;
- 有多个序列存在于白名单中:我们将这个无效barcode改为read支持数量最多的序列;
Linker的处理与Barcode相同。
接头和PolyA序列剪切
在转录组产品中,Read2的末端有可能会出现polyA tail,建库时可能引入的接头序列。我们对这些污染序列进行切除,剪切完的read2长度需要大于设定的最小长度来保证有足够的信息,准确比对到基因组的位置。如果剪切完成后的read2长度小于最小长度,我们认为这条read为无效read。
经过上述处理后,数据组成如下图:
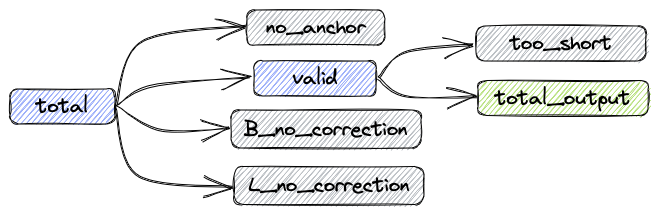
- total: 总共的reads数目
- valid: 不需要矫正和矫正成功的reads数目
- B_corrected: 矫正成功的reads数目
- B_no_correction: 错误Barcode的reads数目
- L_no_correction: 错误Linker的reads数目
- no_anchor:不包含anchor的reads数目
- trimmed: 进行过剪切的reads数目
- too_short: 进行过剪切后长度小于60bp的reads数目
指标之间的关系如下:
total = valid + no_anchor + B_no_correction + L_no_correction
输出fastq的reads数:total_output = valid - too_short
step2: 进行比对并找到比对基因
序列比对
使用STAR比对软件将处理后的R2比对到参考基因组上。
使用QualiMap软件和转录本注释文件GTF,统计reads比对外显子、内含子和基因间区等的比例。
使用featureCounts将比对上的read注释到基因上,可以选择不同的注释规则,如链方向性和定量的feature。当使用外显子定量时,当read超过50%碱基比对到外显子区域时,认为该read来源于此外显子以及外显子对应的基因;当使用转录本定量时,当read超过50%碱基比对到转录本区域时,认为该read来源于此转录本以及转录本对应的基因。
经过上述处理后,有如下数据指标:
- Reads Mapped to Genome: 比对到参考基因组上的reads占所有reads的比例(包括只有一个比对位置和多个比对位置的reads)
- Reads Mapped Confidently to Genome: 在参考基因组上只有一个比对位置的reads占所有reads的比例
- Reads Mapped to Intergenic Regions:比对到基因间隔区reads占所有reads的比例
- Reads Mapped to Intronic Regions:比对到内含子reads占所有reads的比例
- Reads Mapped to Exonic Regions:比对到外显子reads占所有reads的比例
step3: 定量
UMI定量
SeekSoulTools以barcode为单位提取featureCounts输出的bam数据,统计注释到基因的UMI和UMI对应的reads数:
- 过滤掉对应UMI为单个重复碱基的reads, 例如UMI为TTTTTTTT
- 当read注释到多个基因,在有唯一外显子的注释时,为有效read,其他情形下都过滤掉
UMI矫正
测序过程中,UMI也有一定概几率出现测序错误。SeekSoulTools默认使用UMI-tools的adjacency方法对UMI进行矫正。
 图片来源: https://umi-tools.readthedocs.io/en/latest/the_methods.html
图片来源: https://umi-tools.readthedocs.io/en/latest/the_methods.html
细胞判定
在一个细胞群中,我们认为细胞和细胞的mRNA的含量不会相差太多。如果一个barcode对应的mRNA的含量很低,我们认为这个barcode的磁珠并没有捕获细胞,mRNA来源于背景。SeekSoulTools会以上面的规则,进行barcode是否为细胞的判定。有以下几个步骤:
- 对所有barcode按照对应的UMI数由高到低排序;
- 取预估细胞的1%位置的barcode的UMI数除以10为阈值;
- barcode的UMI数大于阈值的判定为细胞;
- barocde的UMI小于阈值,但大于300时,使用DropletUtils分析。DropletUtils方法先假设UMI数量低于100的barcode为empty droplet,然后根据每个droplet相同基因的UMI数总和为背景RNA表达谱中该基因UMI数目,进而得到基因UMI数目的期望值。再通过将每个barcode的UMI数进行统计学检验,显著差异的为细胞;
- 不符合上述条件的为背景。
经过上述处理后,有如下数据指标:
- Estimated Number of Cells: 算法判定的细胞总数
- Fraction Reads in Cells: 判定为细胞的reads占所有参与定量的reads的比例
- Mean Reads per Cell: 细胞的平均reads数,总reads数/判定的细胞数
- Median Genes per Cell: 判定为细胞的barcode中基因数的中位数
- Median UMI Counts per Cell: 判定为细胞的barcode中UMI数的中位数
- Total Genes Detected: 所有细胞检测到基因数量
- Sequencing Saturation: 饱和度,1 - UMI总数/reads总数
step4: 后续分析
经过上述定量,得到表达矩阵后,我们可以进行下一步的分析。
Seurat分析流程
使用Seurat计算线粒体含量,细胞中UMI总数,细胞中基因总数。之后对矩阵进行归一化、寻找高变基因、降维聚类之后寻找差异基因。
fast模块参考fast模块
vdj模块
数据准备
测试数据下载
测试数据 - md5: afbd2deac59b581a4d44a0f73655d71d(物种:人)
wget -c -O PBMC_xin.tar "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/demodata/PBMC_xin.tar"
# decompress
tar xf PBMC.tarcurl -C - -o PBMC_xin.tar "https://seekgene-public.oss-cn-beijing.aliyuncs.com/software/data/demodata/PBMC_xin.tar"
# decompress
tar xf PBMC.tar运行
运行示例
示例1:T细胞受体分析示例
seeksoultools vdj run \
--fq1 /path/to/demo_tcr/demo_tcr_R1.fq.gz \
--fq2 /path/to/demo_tcr/demo_tcr_R2.fq.gz \
--chemistry DD5V1 \
--samplename demo_tcr \
--chain TR \
--core 16 \
--outdir /path/to/ouput/demo_tcr \
--organism human示例2:B细胞受体分析示例
seeksoultools vdj run \
--fq1 /path/to/demo_bcr/demo_bcr_R1.fq.gz \
--fq2 /path/to/demo_bcr/demo_bcr_R2.fq.gz \
--chemistry DD5V1 \
--samplename demo_bcr \
--chain IG \
--core 16 \
--outdir /path/to/ouput/demo_bcr \
--organism human软件参数说明
| 参数 | 参数说明 |
|---|---|
| --fq1 | R1 fastq数据路径。 |
| --fq2 | R2 fastq数据路径。 |
| --samplename | 样本名称。仅支持数字,字母和下划线。 |
| --chemistry | 试剂类型,每种对应一组--shift、--pattern、 --structure、--barcode和--sc5p的组合,可选值:DD5V1;DD5V1 对应SeekOne(R) DD单细胞5'转录组试剂盒。 |
| --organism | 物种,可选值:human,mouse, monkey,rabbit,rat。 |
| --chain | 链类型,可选值:IG,TR; IG对应B细胞受体; TR对应T细胞受体。 |
| --core | 分析使用的线程数。 |
| --outdir | 结果输出目录,绝对路径,并且保证每个任务的outdir唯一。 |
| --skip_misB | barcode不允许碱基错配,默认允许一个碱基错配。 |
| --skip_misL | linker不允许碱基错配,默认允许一个碱基错配。 |
| --skip_multi | 舍弃能纠错为多个白名单barocde的reads,默认纠错为比例最高的barcode。 |
结果文件说明
以下是输出目录结构:每一行代表一个文件或文件夹,用 "├──" 表示。
outs/
├── airr_rearrangement.csv airr格式的结果文件
├── all_contig_annotations.csv 组装得到的所有contig的注释结果文件
├── all_contig.fasta 组装得到的所有contig的fasta格式序列文件
├── clonotypes.csv clonotype相关文件
├── consensus_annotations.csv 以克隆型的链为单位得到一致性序列的注释结果文件
├── consensus.fasta 以克隆型的链为单位得到一致性序列的fasta格式文件
├── filtered_contig_annotations.csv 过滤后的contig的注释结果文件
├── filtered_contig_igblast.fasta 过滤后的contig的fasta格式序列文件
├── metrics_summary.csv 质控指标相关的总结文件
└── report.html 报告文件处理过程
barcode/UMI提取
SeekSoulTools根据Read1的结构设计和参数对barcode/UMI进行提取和处理,对Read1和Read2进行过滤,输出新的fastq文件。
结构设计和描述
barcode和UMI描述 以字母和数字描述Read1的基本结构,字母描述碱基含义,数字描述碱基长度。
B: barcode部分碱基
U: UMI部分碱基
X: 其他任意碱基,用于占位
免疫分析产品的Read1结构为B17U12:
数据流程
Barcode识别和矫正:
根据结构设计,确定barcode所在序列位置,取出相应序列。当取出的barcode序列在白名单中时,我们认为它是有效barcode,计入有效barcode的reads数量;当barcode不在白名单中时,我们认为它是无效barcode。
测序过程中,有一定几率发生测序错误。在提供有白名单的情况下,SeekSoulTools可以尝试barcode矫正。在启用矫正时,当无效barcode一个碱基错配(一个hamming distance)的序列存在于白名单中:
- 只有唯一一个序列存在于白名单中:我们将这个无效barcode矫正为白名单中barcode;
- 有多个序列存在于白名单中:我们将这个无效barcode改为read支持数量最多的序列;
接头和PolyA序列剪切:
在建库时可能引入的接头或者TSO等序列,我们对这些污染序列进行切除,剪切完的read1和read2长度需要大于设定的最小长度来保证有足够的信息,准确比对到基因组的位置。如果剪切完成后的read1或read2长度小于最小长度,我们认为这条read为无效read。
经过上述处理后,数据指标包括:
- total: 总共的reads数目
- valid: 不需要矫正和矫正成功的reads数目
- B_corrected: 矫正成功的reads数目
- B_no_correction: 错误Barcode的reads数目
- trimmed: 进行过剪切的reads数目
- too_short: 进行过剪切后长度小于60bp的reads数目
输出fastq的reads数:total_output = valid - too_short
序列组装
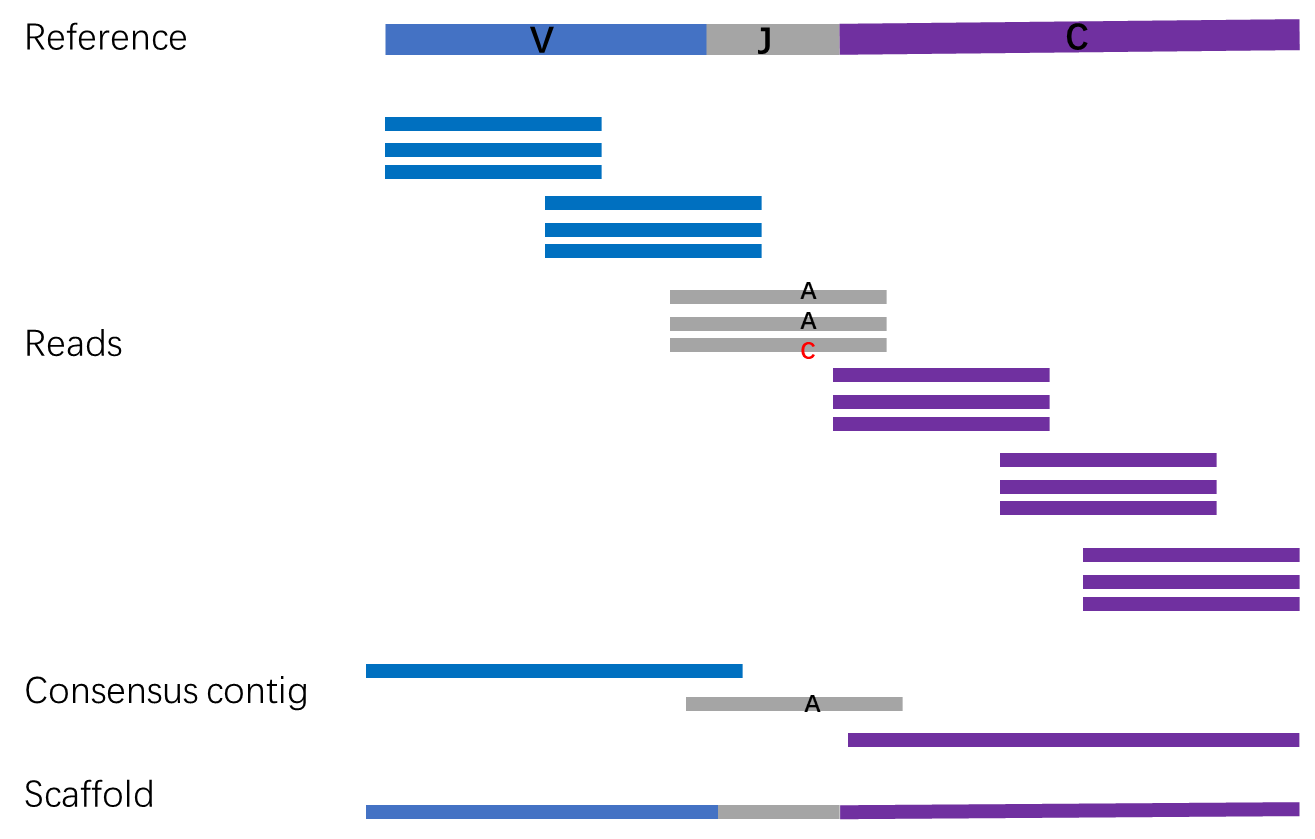
vdj序列组装指的是将测序reads装成为长的vdj序列,主要包含以下步骤(示意图如上):
将reads比对到参考序列,获得相对于参考序列的坐标。
首先,将read和VDJ的germline参考序列分别切成11bp的kmer,然后计算read和参考序列间共有的kmer数目,作为read与参考序列比对长度。接着,如果read和参考序列之间有22bp的碱基是完全匹配的,就认为该read可以比对到参考序列上。对于上述得到的可以比对到germline参考序列的一条read,我们根据比对长度,确认其参考序列是V-REGION,J-REGION或者C-REGION中的哪一种。一条read可以比对到V-REGION(J-REGION或者C-REGION)的多个基因。我们将同一barcode和同一umi的所有reads比对到最多的V-REGION(J-REGION或者C-REGION)的基因,作为最终的参考序列。最后,根据该参考序列,将read上的每个碱基,标记上相对于参考序列的坐标。
对具有相同参考序列坐标的reads上的碱基进行纠错。
分别计算对应于参考序列每个位置上的reads碱基的一致性。如果reads的覆盖深度大于或等于2,并且碱基的一致率大于或等于51%,则选取出现次数最多的碱基;反之则标记为N。
纠错后的reads,组装成长的consensus contig。
使用相对于V-REGION,J-REGION或者C-REGION的坐标,将reads之间进行连接,组装成长的consensus contig。 组装consensus contig的方法有两种(默认为使用第二种方式): 第一种,使用具有同一umi的reads进行组装,每个umi得到一个V-REGION,J-REGION或者C-REGION的contig; 第二种,将同一V-REGION(J-REGION或者C-REGION)基因的所有reads进行合并,分别得到一个V-REGION,J-REGION或者C-REGION的contig。
V-REGION,J-REGION和C-REGION的consensus contig之间的组装。
V-REGION,J-REGION和C-REGION的consensus contig之间通过overlap进行连接,形成完整的序列。将相互之间具有18 bp以上overlap的contig,进行连接。
组装序列过滤
根据组装指标进行过滤:
对于在barcode中同时存在轻链和重链的contig,则符合以下条件之一的contig会被丢掉:对于BCR,如果contig的umi小于3;对于TCR则不做该过滤。
理论上,在一个样本中,与同一种重链所匹配的轻链数量是有限的。而如果存在污染,则会出现一种重链对应很多种轻链的情况。对于umi小于或者等于10的contig,如果符合两个条件之一,会被过滤掉:条件1,在所有barcode中,与重链配对的轻链类型小于10种,并且平均umi数目小于或者等于5,则该轻链及其配对的重链类型。条件2,如果与重链配对的轻链大于10种,并且占比小于50%的轻链及其配对的重链类型。
对于在barcode中仅存在轻链或者重链的contig,则符合以下条件之一的contig会被丢掉:对于BCR,如果contig的umi小于3;对于TCR则不做该过滤。
contig所在的barcode的总reads数目小于或者等于50,则该barcode中的所有contig均丢掉。
对于在多个barcode中出现,并且在超过75%的barcode中都只包含轻链或者重链的,同时umi小于5或者reads小于所有barcodereads数中值1/2的contig。
根据注释结果进行过滤:
根据igblast的结果,仅保留序列全长大于或等于300 bp,并且complete_vdj和productive均为TRUE的contig。
对一个barcode有过多的contig的进行过滤: 对于每个细胞/barcode中chain(例如TRA或者TRB)来讲,最多保留两个序列。如果超出两个,则仅保留出现次数最多的两个CDR3核酸序列对应的组装序列。如果每个CDR3核酸对应多个组装序列,则选取umis和reads数目最多的组装序列。
VDJ注释
注释使用igblast软件,详细请参考https://ncbi.github.io/igblast/。
参考序列下载子IMGT网站(http://www.imgt.org),包含人,猴,小鼠,大鼠和兔等五个物种。
细胞鉴定
经组装序列过滤后,包含一个及以上contig的barcode,判定为细胞。
clonotype计算
clonotype计算使用changeo,详细请参考https://changeo.readthedocs.io/en/stable/。
utils模块
addtag
对bam进行修改,添加barcode和umi标签。
运行示例
为分析设置必要的配置文件,包括样本的bam文件和step3目录下的umi.xls文件。使用以下命令运行SeekSoulTools:
seeksoultools utils addtag \
--inbam step2/featureCounts/Samplename_SortedByName.bam \
--umifile step3/umi.xls \
--outbam Samplename_addtag.bam软件参数说明
| 参数 | 参数说明 |
|---|---|
| --inbam | 输入的bam,step2 featureCount目录下的bam文件。 |
| --outbam | addtag之后的bam文件名称 |
| --umifile | 输入的umi文件,seeksoultools输出目录step3的umi.xls文件 |
常见问题
如何构建参考基因组?
参考基因组的构建请参考如何构建参考基因组?。
如何选择chemistry?
chemistry参数的设定跟所用试剂类型相关,比如全序列和FFPE产品选择DD-Q,DD单细胞3'转录组试剂盒请选择DD—V2, DD单细胞5'转录组试剂盒选择DD5V1等。如果chemistry参数选择错误可直接在log日志中查找"valid barcode rate of"相关信息,后面的比例太低则表示chemistry不正确。或者在samplename_summary.json文件中可查看valid 和total的比例。
为什么出现FASTQ相关报错?
软件报错"Error while decompressing extra concatenatedgzip files on *fastq.gz\n"? 根据提示,输出的fq文件应该是不完成的,请核对参数–fq1 --fq2参数的文件是否完整。同样是fq数据不完整,也可能出现如下错误信息"FileFormatError: Error in sequence file at unknown line: Reads are improperly paired. There are more reads in file 1 than in file 2." 或者"Error in FASTQ file at line 4: Premature end of file encountered. The incomplete final record was: '@A01565:134:HKFGNDSX3:1:1101:11080:2957 1:N:0:CACTTCGA+ACAGCTGC\nCCATCACTACGGAAGGTTGAGCTCTATGATTTTTT…ATTATTATT\n+\n' err!!!",总而言之,请一定确保自己的fq文件是完整且配对的。
为什么没有barcodes.tsv.gz文件?
软件报错"No such file or directory: '/outputpath/samplename/step3/filtered_feature_bc_matrix/barcodes.tsv.gz'" ? 首先查看输出目录,如果有raw_feature_bc_matrix,并且该目录下的三个文件大小没有异常小,此时可以单独运行一个细胞判定的脚本Rscript /path/seeksoultools/lib/python3.XX/site-packages/seeksoultools/utils/cell_identify.R -i outputpath/step3/raw_feature_bc_matrix -e 3000(该命令在日志中有输出) ,当执行此脚本时应该会出现报错信息,比如缺少某个R包的问题,此时请确认,1.2.1版本之前的,在脚本执行的时候先激活环境,比如请执行 source activate seeksoultools,或者export PATH=/path/seeksoultools/bin:$PATH,激活环境后重新运行脚本。另外温馨提示,在安装seeksoultools软件时不要把它软链到另一个磁盘等,请直接安装,并且保证环境名称是唯一的,在运行脚本的时候使用绝对路径。
如果检查输出目录中raw_feature_bc_matrix目录下的文件大小异常小,比如只有几个字节大小,此时可能是gtf文件有问题。具体的gtf格式说明及要求,可以参考如何构建参考基因组?。
为什么出现samtools报错?
软件报错"stderr: samtools sort: failed to read header from '/outputpath/samplename/.test/test_1M_Aligned.out.bam'"? 根据提示bam文件有问题,此时请查看/outputpath/samplename/.test/test_1M_Log.out。文件可能会提示'FATAL INPUT ERROR: unrecognized parameter name "genomeType" in input "genomeParameters.txt"',或者'EXITING because of FATAL ERROR: Genome version: 2.7.1a is INCOMPATIBLE with running STAR version: 2.7.10a SOLUTION: please re-generate genome from scratch with running version of STAR, or with version: 2.7.4a'也会提示索引的版本和STAR软件的版本。报错原因是二者版本不一致,请给软件传递参数–star_path 给定构建索引用的STAR软件路径。
用1.2.2版本seeksoultools分析完数据得到的web sumarry中线粒体比例都是0? seeksoultools是根据gtf中gene_name的value中是否以Mt- 或者mt- 开头来判断线粒体基因的,如果gtf中gene_name的value没有进行这种修改就会导致报告中mt比例均为0.
为什么没有rnaseq_qc_results.txt?
软件报错"No such file or directory: '/outputpath/samplename/step2/STAR/rnaseq_qc_results.txt'" 根据提示,软件在qualimap步骤运行失败,对于1.2.1及其之前的版本,需要在下载完软件包之后进行unpack。具体操作方式为:"source ./bin/activate ; ./bin/conda-unpack"
为什么出现biotype的问题?
软件运行报错"gene_biotype = re.search(r'(gene_type|gene_biotype|transcript_biotype|transcript_type) "(.*?)";', ids).group(2) AttributeError: 'NoneType' object has no attribute 'group'" 在1.2.1及其之前版本fast模块要求gtf文件的attribute列中有记录gene的biotype,当出现这个提示的时候,可以修改gtf文件,确保gene有biotype,或者可以用1.2.2及其之后的版本,当读取不到基因的biotype是,默认type为"undefine"。
关注的某个基因,最后矩阵中为什么表达量都是0?
在进行定量的时候,如果某条reads即比对到A基因又比对到B基因,在1.2.0之前的版本是直接丢掉,1.2.0及其之后的版本会对基因的位置做进一步判定,如果只有某个基因比对到的是exon区间,其他基因比对的是非exon区间,则将该reads判定给该基因。进行排查时,首先从step2/featureCounts/*_SortedByName.bam里面提取出'XT'tag中包含该基因(ensemble id 而非symbol)的reads,然后针对这些reads逐条检查,是不是XTtag中包含的多个基因,且目标基因比对的区域非exon或者XTtag包含的基因中有多个是比对的exon区域。
发布说明
v1.2.2
- 添加vdj分析模块
- fast模块对FFPE样本的支持
- 修复gtf中没有lncRNA类型引起基因中位数的统计错误
- 解决基因名称重复导致差异基因缺失EnsemblID的问题
- 其他改进,提升易用性
v1.2.1
- 更新报告样式
- 增加SP1、SP2和TSO和接头的剪切
- 增加对bam添加addtag的工具
- 增强对非标准gtf的支持
- fast模块支持人和鼠之外的物种
v1.2.0
- 增加去除barcode和umi序列后的read1的fq文件输出
- 添加fast分析模块
- 应用UMI-tools的矫正方法
- 更新read注释到多个基因时的处理规则:在有唯一外显子的注释时,视为有效read
v1.0.0
- 首次发布
